Generación de árboles filogenéticos UPGMA a partir de un código de proteína, en Python
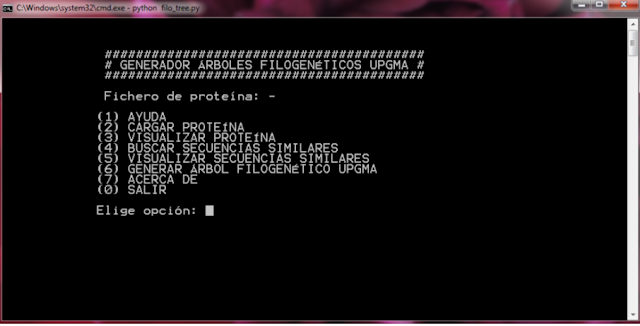
En este post se presenta una aplicación para la generación de árboles filogenéticos UPGMA a partir de un código de proteína en formato FASTA . La aplicación se puede descargar de: https://sites.google.com/site/elviajedelnavegante/codigo/FILO_TREE.zip?attredirects=0&d=1 Los árboles filogenéticos son útiles para encontrar los familiares más cercanos a nuestro organismo de estudio. Se ha implementado mediante la creación de un script Python , que utiliza la biblioteca externa BioPython para la creación de árboles filogenéticos. La aplicación permite obtener secuencias similares a partir de una secuencia de proteína dada. Estas secuencias obtenidas por Blast son procesadas con un algoritmo de alineamiento múltiple ( Clustal ), cuyo resultado sirve para crear la matriz de distancias y el árbol filogenético UPGMA. La aplicación es multiplataforma, para sistemas Microsoft Windows y Linux . La configuración permite seleccionar cuántas secuencias como máximo se quiere u...